 Az EpigenPlot egy metilációs adatok összehasonlítására alkalmas platform egy általunk létrehozott adatbázis alapján. Az adatbázis több tanulmány nyilvánosan elérhető adatára épül. Az összes tanulmány esetén valamelyik Illumina microarray: az Infinium HumanMethylation450K, vagy az Infinium HumanMethylationEPIC platformot alkalmazták és kezeletlen személyek normál és/vagy tumoros szöveteit vizsgálták. Az EpigenPlot jelenleg vastagbélszövetek (normál, adenoma, adenokarcinoma) tanulmányozására alkalmas 2346 minta alapján.
Az EpigenPlot egy metilációs adatok összehasonlítására alkalmas platform egy általunk létrehozott adatbázis alapján. Az adatbázis több tanulmány nyilvánosan elérhető adatára épül. Az összes tanulmány esetén valamelyik Illumina microarray: az Infinium HumanMethylation450K, vagy az Infinium HumanMethylationEPIC platformot alkalmazták és kezeletlen személyek normál és/vagy tumoros szöveteit vizsgálták. Az EpigenPlot jelenleg vastagbélszövetek (normál, adenoma, adenokarcinoma) tanulmányozására alkalmas 2346 minta alapján.
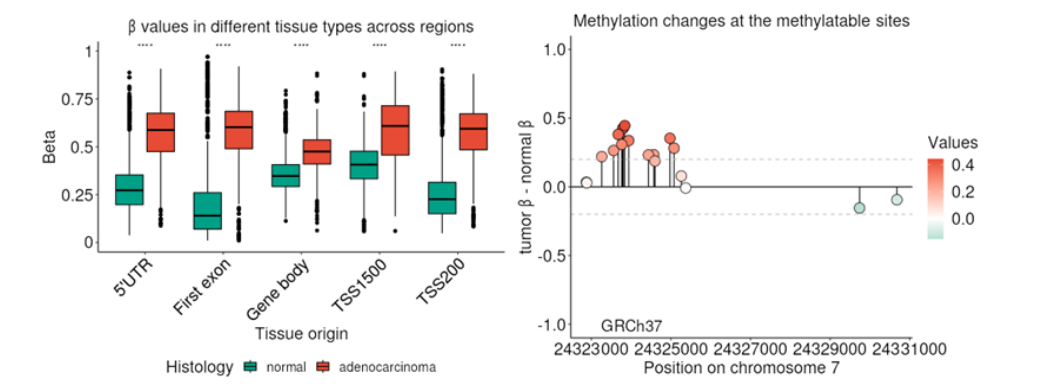
Az összehasonlítás elsősorban gén alapú, nem tartalmazza a genom intergénikus régióinak vizsgálatát. Négy fő funkciója van: gén, gén régió (pl. első exon, 5’UTR), CpG (egy génen belül) és KEGG-útvonal szinten lehet elemzéseket végezni. A felhasználó a vizsgálandó gén metilációját szövettípusonként (normál, tumor) összehasonlítva, illetve gén régiókra lebontva ábrázolhatja, ami lehetőséget ad a lehetséges biomarkerek validálására.
A CpG szintű elemzés esetén a gén területén vizsgált összes CpG ábrázolásra kerül és interaktív módon kiválasztható a megcélzandó lókusz, amiről így több információ szerezhető. Mindemellett ez a funkció alkalmas a CpG szigetek metilációjának szemléltetésére is. Nagy méretű és/vagy sok CpG lókuszt tartalmazó gén esetén lehetőség van az ábrázolt régió leszűkítésére is.
A KEGG útvonal szintű elemzés egy adott útvonalhoz tartozó tetszőleges számú (n) gén ábrázolására alkalmas. A gének a metiláció változás mértéke szerint rangsorolva vannak és az ennek a sornak megfelelő első n gén kerül ábrázolásra. Ezen kívül az útvonal összes génje megtekinthető egy táblázatban.
Minden elemzés esetén tetszőleges összeállítású adatszett (pl. csak ugyanabból a személyből származó normál-tumor párokat tartalmazó), illetve platform (HM450K vagy EPIC) választható. Terveink szerint az EpigenPlot a jövőben kibővül emlőrák adatokkal is.
Elérhetőség: https://www.epigenplot.com